|
|
|
|
|
QB期刊 | 基于UMI短读长数据集分析纠错如何影响PCR去重 |
|
|
论文标题: How error correction affects polymerase chain reaction deduplication: A survey based on unique molecular identifier datasets of short reads
期刊:Quantitative Biology
作者:Pengyao Ping, Tian Lan, Shuquan Su, Wei Liu, Jinyan Li
发表时间:21 Apr 2025
DOI:10.1002/qub2.99
微信链接:点击此处阅读微信文章
下一代测序(NGS)数据已广泛应用于生物信息学的各类下游分析。为消除测序过程中引入的偏差与错误,科研人员开发了众多PCR去重与纠错技术。然而,针对短读长的PCR去重与纠错进行联合分析的研究尚属空白,尤其缺乏利用UMI数据集评估纯计算方法性能、探究纠错对去重影响的系统性研究。
近日,悉尼科技大学/中国科学院深圳先进技术研究院李金艳教授团队在 Quantitative Biology 期刊发表综述文章"How error correction affects polymerase chain reaction deduplication: A survey based on unique molecular identifier datasets of short reads"。该文不仅全面梳理了短读长PCR去重与纠错领域的最新进展,更通过严谨的比较分析揭示了一个严峻现实:现有纯计算工具虽能消除部分错误,但仍遗留数十万个错误读段未被校正,甚至引入大量新序列,对PCR去重毫无裨益。基于这些发现,文章指明了未来研究方向,为改进计算方法、提升短读长测序质量提供了重要思路。

全文概要
下一代测序(NGS)技术以其前所未有的速度、精度和成本效益,彻底革新了基因组学研究。文库构建与簇扩增阶段的PCR扩增是NGS关键步骤,用于增加DNA/RNA分子片段数量以确保可靠测序。然而,PCR扩增存在固有偏好性,可能产生嵌合分子,其错误分布极为复杂。这些扩增偏差与错误模式严重干扰微生物组分析、变异检测、RNA定量等下游应用。为此,学界已提出多种生物学方法与计算学方法用于PCR去重。
除PCR相关错误外,样本处理和碱基判读(如荧光串扰)等环节也会引入测序错误,进一步削弱数据分析的准确性。因此,克服PCR及其他步骤的偏差与错误对保障NGS数据可靠性至关重要。NGS纠错技术的核心目标是识别并修正fasta/fastq数据集中由PCR或碱基判读导致的错误碱基。图1展示了NGS中分子追踪、PCR去重与纠错的全过程概览。
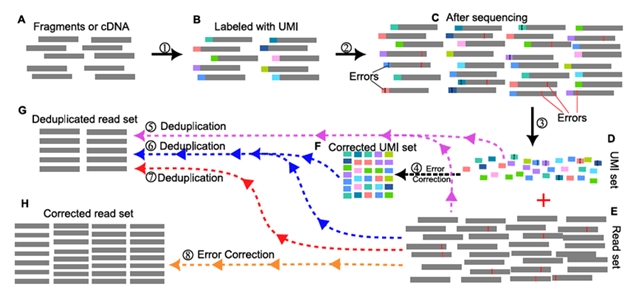
图1. 分子追踪、PCR去重和纠错过程的示意图概览。(A–C) 展示了UMIs在精确分子追踪中的应用,该过程考虑了测序过程中引入的错误,这些错误可能同时出现在UMI序列和读段序列中。(D–G) 描述了PCR去重过程:⑤表示使用UMI标签去除重复读段,而④-F-⑥描绘了使用校正后的UMI标签消除重复的过程。⑦代表仅使用计算方法去除重复读段。(E−⑧−H) 阐明了旨在消除测序过程中引入的所有错误的计算纠错过程。值得注意的是,去重后的读段集合因所采用的PCR去重过程而异,而纠错方法的过度校正可能会无意中去除真实的原始读段。
短读长PCR去重研究进展
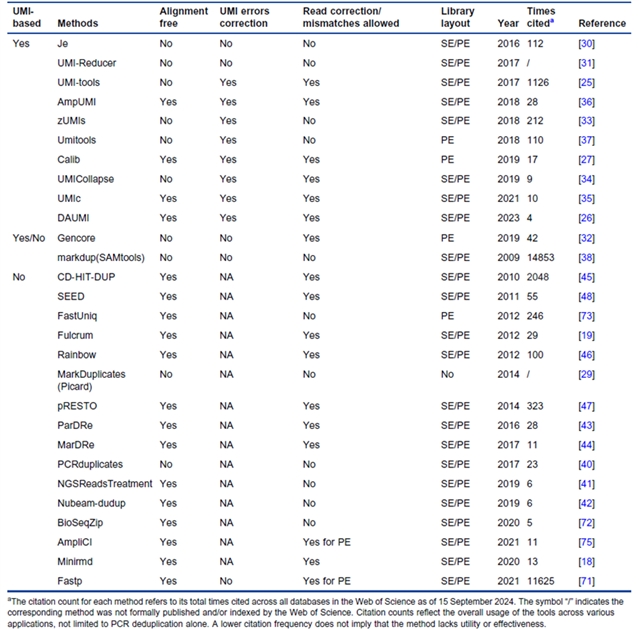
现有PCR去重方法分为两类:一类利用UMI等生化技术精确追踪分子;另一类仅依赖纯计算。两类方法均可按是否使用参考基因组进一步细分(表1)。
表1. PCR去重方法汇总
纠错方法研究进展
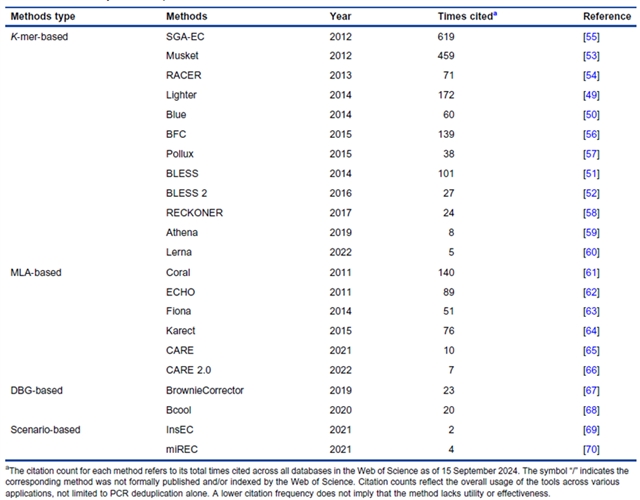
纠错算法采用统计/数学模型检测并修正错误以提升数据质量。现有方法均为不依赖UMI的计算方法,可分为k-mer法、多序列比对(MSA)法和de Bruijn图(DBG)法三类,另有一些针对特定场景的专用方法,如表2所示。
表2. 纠错方法汇总
PCR去重方法及纠错方法的多维度性能测试
研究选取11个UMI单端数据集和1个配对末端数据集,系统评估了多种PCR去重方法及纠错方法的综合表现,主要发现如下:
1. PCR去重方法性能比较
1)在11个单端数据集上,NGSReadsTreatment、Nubeam-dedup、BioSeqZip和Fastp四种纯计算方法的去重结果与原始数据集几乎完全一致,说明这些方法仅识别唯一读段,未纠正PCR偏差与错误。
2)三种UMI方法(UMI-tools、AmpUMI、UMIc)使唯一读段数平均减少88.61%、82.97%和88.69%,但UMI-tools和UMIc引入大量新序列(8.24%-20.27%),存在过度纠错。
3)允许错配的工具(ParDRe、Minirmd)可消除部分PCR错误,但错配阈值从0增至3时,唯一读段数持续下降,显示其可能误删真实序列。
4)在配对末端数据集上,多数UMI方法因内存或重建问题失效,而Fastp产生的大量新序列显示其过度纠错倾向。
2. UMI方法间的一致性分析
UMIc与AmpUMI在单端数据集上重叠度较高(86.4%),但UMIc引入的新序列暗示其过度纠错。UMI-tools因依赖参考基因组比对而与其他方法交集较小,表现不稳定。
3. 计算方法与UMI方法的基准对比
AmpUMI的去重结果是所有纯计算方法(允许0错配)的子集。当允许错配时,重叠度下降,证实CD-HIT-DUP、ParDRe、Minirmd可能因过度纠错而扭曲真实读段。
4. 运行效率评估
AmpUMI在UMI方法中速度最快(<1分钟,<550 MB)。多数计算方法在1分钟内完成,但ParDRe耗时超1小时。Minirmd内存效率最佳。
5. 纠错方法性能比较
1)所有纠错方法均引入数万至数十万新序列,且遗留大量错误读段未校正。
2)各方法间唯一读段集合重叠有限,显示结果差异显著,无"通用最优"方法。
3)Coral和Bcool分别在两类数据集上引入新序列最少,与UMI结果重叠度最高。
纠错对PCR去重的影响
将8种纠错方法(BFC、Bcool、Care、Coral、Fiona、Lighter、Pollux和RACER)与3种去重方法(CD-HIT-DUP、ParDRe、Minirmd)组合测试发现:现有纠错方法对PCR去重无益,反而降低与UMI金标准的重叠度。纠错后的去重结果仍远大于UMI去重结果,证实现有方法无法有效还原真实序列。
结论与展望
本研究首次系统揭示了纯计算PCR去重与纠错方法的系统性缺陷:去重结果与UMI金标准差异显著,纠错工具引入大量伪序列,且方法表现高度依赖数据类型。
尽管UMI是缓解不确定性的有效手段,但仍面临挑战:1)UMI自身可能发生PCR错误,难以区分早期PCR错误与真实序列;2)短UMI在大规模测序中易发生碰撞(DAUMI方法可部分解决);3)实验成本高、耗时长,限制大规模应用;4)基于比对的方法依赖参考序列质量,可能误删真实序列。
未来改进的方向有:1)引入筛选机制:结合序列丰度等信息,准确鉴别真实近似重复,避免误删(如miREC方法的成功实践);2)整合工作流程:去重工具应记录ID与丰度信息,纠错工具应输出唯一读段,实现无缝衔接(Calib和DAUMI已有成功尝试);3)深度学习赋能:突破传统k-mer/MSA/DBG框架,结合平台特异性错误模式与机器学习,开发更智能的算法。
本研究为NGS数据处理流程敲响了警钟:唯有正视现有工具的局限性,才能开发出真正可靠、不引入新错误、保留真实序列与丰度信息的理想算法,推动精准医疗与科研进步。
QB期刊介绍
Quantitative Biology (QB)期刊是由清华大学、北京大学、高教出版社联合创办的全英文学术期刊。QB主要刊登生物信息学、计算生物学、系统生物学、理论生物学和合成生物学的最新研究成果和前沿进展,并为生命科学与计算机、数学、物理等交叉研究领域打造一个学术水平高、可读性强、具有全球影响力的交叉学科期刊品牌。
《前沿》系列英文学术期刊
由教育部主管、高等教育出版社主办的《前沿》(Frontiers)系列英文学术期刊,于2006年正式创刊,以网络版和印刷版向全球发行。系列期刊包括基础科学、生命科学、工程技术和人文社会科学四个主题,是我国覆盖学科最广泛的英文学术期刊群,其中12种被SCI收录,其他也被A&HCI、Ei、MEDLINE或相应学科国际权威检索系统收录,具有一定的国际学术影响力。系列期刊采用在线优先出版方式,保证文章以最快速度发表。
中国学术前沿期刊网
http://journal.hep.com.cn

特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。