蛋白质是生命活动的执行者,由20种天然存在的蛋白质氨基酸构成。与高歌猛进的DNA测序技术相比,高灵敏、高通量的蛋白质测序技术的发展严重滞后,目前仅Edman降解法和质谱法能做到蛋白质测序。然而,Edman降解法的序列分析长度有限,质谱法的检测限和动态范围阻碍了低丰度蛋白质的分析鉴定和新型生物标志物的发现。单分子蛋白质测序技术可以提供更高的灵敏度和准确度,有望助力单细胞蛋白质组学的发展。2023年,《Nature》杂志将单分子蛋白质测序技术评为未来一年对科技创新产生巨大影响的七项新技术之一。
相比于核酸的4种核苷酸基本单元,蛋白质由20种氨基酸组成,具有更复杂的化学多样性。无论采取哪种读出方式,检测20个可区分的信号本身就是一个很大的挑战,准确解读蛋白质上的翻译后修饰信息对分析方法的检测分辨率提出了更高的要求。
近日,南京大学化学化工学院黄硕教授课题组构建了一种工程化耻垢分枝杆菌膜蛋白A (MspA) 纳米孔,实现了全部蛋白质氨基酸及它们的翻译后修饰检测。研究团队在孔道的收缩区域引入了一个镍离子-次氮基三乙酸(Ni-NTA)适配体,利用配位相互作用实现了全部蛋白质氨基酸的直接检测和区分,区分准确率高达98.8%,是世界上第一个能完全分辨所有20种蛋白质氨基酸的纳米孔。在本篇工作中,作者亦采用了同种孔道,进而同时区分了4种常见的翻译后修饰氨基酸。

图1:基于Ni-NTA修饰的MspA纳米孔对20种氨基酸的检测
随后,作者将该策略用于多肽的氨基酸组成分析。作者使用氨肽酶将肽消化为单个的氨基酸,然后利用纳米孔读取电流事件,经过机器学习识别氨基酸身份,最终重构出原始肽序列的氨基酸图谱。
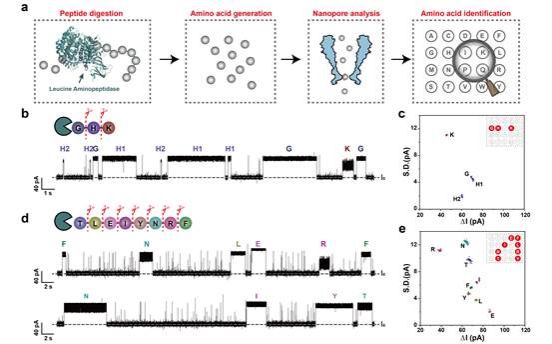
图2:基于纳米孔策略的肽的氨基酸组成鉴定
该纳米孔传感器还可以在分辨20种氨基酸的同时直接分辨翻译后修饰(如磷酸化、甲基化、糖基化、乙酰化),对纳米孔蛋白质测序和翻译后修饰的测序检测具有里程碑意义。
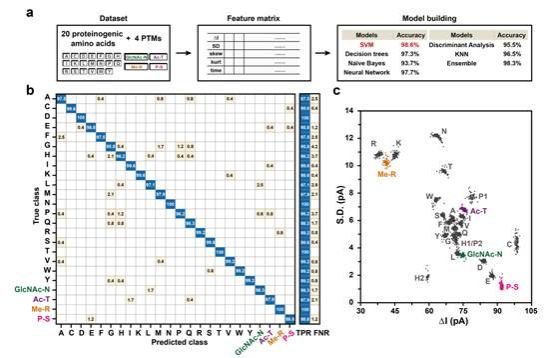
图3:机器学习辅助20种天然氨基酸及4种修饰氨基酸的识别
该工作以“Unambiguous discrimination of all 20 proteinogenic amino acids and their modifications by nanopore”为题,于2023年9月25日在《Nature Methods》发表相关论文(论文链接:https://doi.org/10.1038/s41592-023-02021-8)。南京大学黄硕教授为该论文唯一通讯作者,化学化工学院博士生王可凡、张善雨及物理学院博士生周骁为该论文共同第一作者。此项研究得到了科技部国家重点研发计划、国家自然科学基金、江苏省自然科学基金等经费支持。
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。