|
|
|
|
|
QB期刊封面 |贾耿介/李煜/高欣/Andrey Rzhetsky团队合作提出将医疗记录编码到高维疾病空间中的嵌入方法 |
|
|
论文标题:An effective encoding of human medical conditions in disease space provides a versatile framework for deciphering disease associations
期刊:Quantitative Biology
作者:Tianxin Xu, Yu Li, Xin Gao, Andrey Rzhetsky, Gengjie Jia
发表时间:11 Nov 2024
DOI:10.1002/qub2.93
微信链接:点击此处阅读微信文章
从大规模、异构的健康数据中揭示复杂的疾病关联仍是一项重大挑战。传统统计方法和简单的机器学习算法难以有效整合和分析这类多样化的医疗数据,从而限制了分析的准确性与深度。此外,这些方法通常将疾病视为彼此独立的离散实体,忽略了共病关系、病理通路以及表型重叠等关键联系。
近期,中国农业科学院(深圳)农业基因组研究所贾耿介团队,联合香港中文大学李煜团队、沙特阿卜杜拉国王科技大学高欣团队以及芝加哥大学Andrey Rzhetsky团队,在 Quantitative Biology 期刊发表了一篇题为 “An effective encoding of human medical conditions in disease space provides a versatile framework for deciphering disease associations” 的封面文章。详细介绍了一种将医疗记录编码到高维疾病空间的嵌入方法,可用于揭示疾病关联、实现数据驱动的疾病分类、辅助遗传参数估计,并分析共病的遗传基础。文章还讨论了该方法面临的挑战与未来前景。


全文概要
研究团提出了一种高效的疾病嵌入(disease embedding)方法(图1),将人类疾病编码到高维向量空间中,为系统解析疾病关联提供了多功能计算框架。该方法将稀疏、大规模、多模态的健康数据(如电子健康记录)映射为连续的向量表示,从而量化疾病之间的相似性,并支持多种下游分析,如疾病关联研究和遗传学分析。同时,研究还讨论了医疗文本输入、在线训练、结果验证以及多模态基础模型构建等方面的挑战。
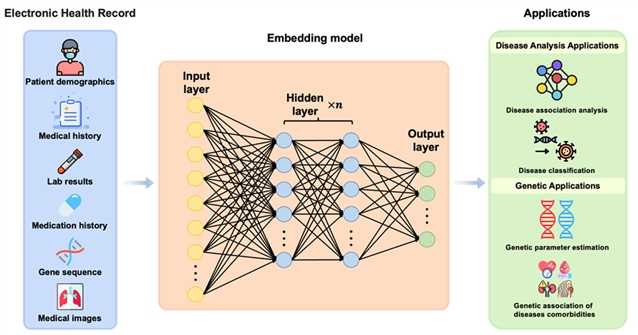
图1. 疾病嵌入工作流
疾病嵌入工作流
从电子健康记录数据收集(包括人口统计、医疗历史、实验室结果、药物历史、基因序列和医疗图像),经预处理后输入嵌入模型(如神经网络),生成高维疾病向量,用于疾病关联分析、疾病分类、遗传参数估计和共病遗传分析等应用。
疾病嵌入在疾病分析中的应用
疾病嵌入技术 (Disease Embedding) 将复杂的疾病条目转换为高维向量,为疾病分析提供了全新的计算范式,其应用主要体现在以下两个方面:
(1)揭示隐性疾病关联与类比。通过在嵌入空间中计算向量相似性(如余弦相似度),研究人员能够量化疾病间的深层联系,挖掘出传统网络分析或机器学习方法难以捕捉的非显性关联。疾病嵌入的方法支持向量代数运算,从而揭示了疾病间的类比关系(例如,A+B ≈ C,如“软骨钙质沉着症 + 结缔组织感染 ≈ 化脓性关节炎”)。在人类表型本体(HPO)数据中,疾病嵌入方法已被证明在表型驱动的疾病诊断上,其效率和准确性均优于传统算法。
(2)构建数据驱动的疾病分类学。将疾病向量应用于聚类算法(如k-means或层次聚类),能够生成一种数据驱动的、新型的疾病分类体系,这挑战了现有的临床分类标准。例如,在ICD-9系统中,嵌入分析揭示了偏头痛(传统上归类于中枢神经系统疾病)与免疫系统疾病之间存在更强的关联性。
疾病嵌入在遗传学研究中的应用
疾病嵌入为遗传学研究,特别是复杂性状和共病 (Comorbidity) 研究,开辟了新的途径:
(1)辅助遗传参数估计。估算遗传力 (heritability) 和遗传相关性 (genetic correlation) 等关键参数,传统上严重依赖于大规模遗传、家系或GWAS汇总数据(例如LDSC方法)。疾病嵌入技术代表了一个范式转变,它允许在缺乏此类遗传数据的情况下,通过计算模型预测这些参数。该方法在预测遗传力方面表现出高准确性,其预测的遗传相关性与LDSC等方法得出的结果高度一致。
(2)识别共病的遗传基础。该方法将疾病嵌入的向量坐标视为一种新型的量化表型。研究人员可以利用这些新型表型特征进行全基因组关联研究 (GWAS),从而识别出与疾病共病模式相关的特定遗传关联。这为理解多种疾病并发的生物学机制提供了有力工具。
挑战与展望
尽管疾病嵌入在揭示疾病关系方面展现出巨大潜力,但这一方法仍面临严峻的挑战。模型输入方面,存在数据集成困难(如ICD-9与ICD-10的标准化)、数据缺失和偏见,以及医学术语的多义性(polysemy)和同义性(synonymy)问题。模型能力方面,迫切需要能适应流式数据的在线训练算法,以及能整合图像、基因组学等信息的多模态基础模型。结果验证方面,必须确保计算结果反映的是真实的生物学现象而非数学伪影,进行包括专家审查、临床关联性分析以及体内和体外实验在内的严格验证。
在疾病关联研究中,疾病嵌入模型为捕捉异构、多模态健康数据中的复杂关系提供了强大框架。通过将疾病表示为向量,它不仅有助于理解疾病间的潜在联系,也为更精准的医疗决策提供支持。未来,疾病嵌入有望与实时临床决策系统结合,用于识别共病与疾病进展,实现个体化护理。随着多组学和纵向电子健康记录数据的不断丰富,嵌入模型可进一步整合蛋白质组、代谢组及表观组学信息,更准确地刻画疾病机制与时间动态,推动精准医学的发展。

QB期刊介绍
Quantitative Biology (QB)期刊是由清华大学、北京大学、高教出版社联合创办的全英文学术期刊。QB主要刊登生物信息学、计算生物学、系统生物学、理论生物学和合成生物学的最新研究成果和前沿进展,并为生命科学与计算机、数学、物理等交叉研究领域打造一个学术水平高、可读性强、具有全球影响力的交叉学科期刊品牌。
《前沿》系列英文学术期刊
由教育部主管、高等教育出版社主办的《前沿》(Frontiers)系列英文学术期刊,于2006年正式创刊,以网络版和印刷版向全球发行。系列期刊包括基础科学、生命科学、工程技术和人文社会科学四个主题,是我国覆盖学科最广泛的英文学术期刊群,其中12种被SCI收录,其他也被A&HCI、Ei、MEDLINE或相应学科国际权威检索系统收录,具有一定的国际学术影响力。系列期刊采用在线优先出版方式,保证文章以最快速度发表。
中国学术前沿期刊网
http://journal.hep.com.cn

特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。