|
|
|
|
|
QB 北京大学席瑞斌团队提出单细胞转录组混合细胞类型的基因调控网络推断框架 |
|
|
论文标题: Single-cell gene regulatory network analysis for mixed cell populations
期刊:Quantitative Biology
作者:Junjie Tang, Changhu Wang, Feiyi Xiao, Ruibin Xi
发表时间:02 July 2024
DOI:https://doi.org/10.1002/qub2.64
微信链接:点击此处阅读微信文章
基因调控网络(Gene Regulatory Network, GRN)描述了基因之间的调控关系,对于解析复杂生物系统具有重要意义。传统上,GRN的推断主要基于bulk RNA测序数据。然而,由于bulk RNA测序数据源自混合的细胞群体,仅能反映群体平均的基因表达水平,难以揭示单细胞层面的调控特性。近年来,单细胞RNA测序(scRNA-seq)技术的兴起,为在单细胞分辨率下研究基因表达和推断GRN提供了前所未有的机遇。针对scRNA-seq数据的特点,研究者们开发了多种GRN推断方法,这些方法通常假设所有细胞共享相同的GRN。然而在实际情况中,scRNA-seq数据往往包含多种细胞类型,而每种类型通常具有特定的GRN和独特的基因表达模式。由于单细胞的细胞类型通常未知,传统方法采用两步流程来估计GRN:首先对单细胞进行聚类分类以确定细胞类型,然后基于分类结果推断各细胞类型的GRN。这种方法在细胞类型间差异显著且分离良好的情况下效果较佳,但当细胞类型高度混杂时,大量细胞的类型无法准确分配,导致GRN推断的准确性显著下降。
近期,北京大学统计科学中心席瑞斌团队在Quantitative Biology期刊上发表了一篇题为“Single-cell gene regulatory network analysis for mixed cell populations”的研究论文,提出了一种基于混合柏松对数正态分布对scRNA-seq数据建模的新方法VMPLN。该方法能够在混合细胞群体中同时进行聚类和网络推断,从而显著提升细胞类型特异性GRN推断的准确性和鲁棒性。

全文概要
为了推断 scRNA-seq 数据中混合群体的基因调控网络(GRN),研究团队采用混合泊松对数正态 (MPLN) 模型对混合群体的 scRNA‐seq 计数表达数据进行建模,建立了VMPLN方法。该方法能在模拟中准确恢复了不同细胞群体的基因网络和scRNA-seq 基准数据中的基因调控关系,并在COVID-19 患者的 scRNA-seq 数据中揭示了关键的生物学过程。VMPLN 方法通过联合聚类与网络推断,为单细胞 RNA 测序(scRNA-seq)数据中的基因调控网络(GRN)推断提供了一种高效且精准的工具。
VMPLN 框架
VMPLN 框架采用了混合泊松对数正态 (MPLN) 模型来描述带混合群体的scRNA-seq计数表达数据的生成机制(如下面公式所示):
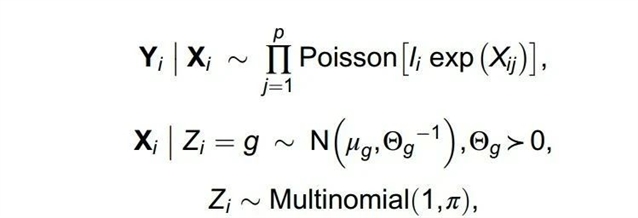
进一步,由于基因调控网络通常具有稀疏性特征,研究团队通过最小化以下带有稀疏性惩罚的负对数似然函数来优化模型参数(如下面公式):
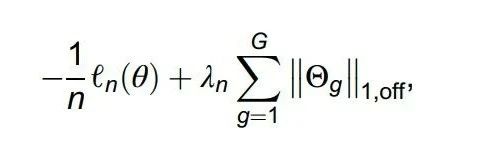
然而,由于混合泊松对数正态 (MPLN)的对数似然函数涉及复杂的积分,直接最小化该对数似然函数具有较大挑战性。因此,研究团队采用变分推断方法来估计细胞特异性GRN的精度矩阵 。大致来说,通过计算Evidence Low BOund (ELBO) 函数来近似该对数似然函数,并通过最小化带惩罚的ELBO函数来估计模型参数(如下面公式所示):
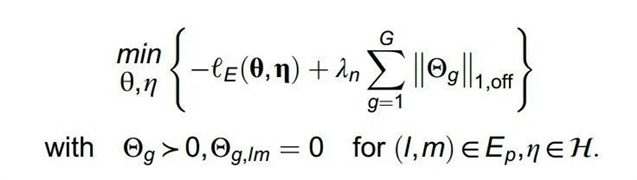
通过最小化综合完全似然准则 (Integrated Complete Likelihood, ICL),为每个细胞类型特异的基因调控网络选择合适的超参数(如下面公式所示):
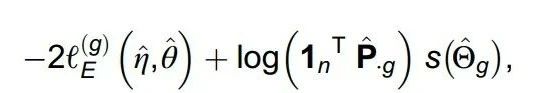
VMPLN 在模拟中准确恢复了不同细胞群体的基因网络
研究团队利用 MPLN 模型生成了包含多个细胞类型的模拟 scRNA-seq 计数表达数据,以评估 VMPLN 在估计细胞特异性基因调控网络方面的性能,并与现有的图模型方法和单细胞 GRN 推断方法进行了比较。图1展示了在不同场景下,各算法 pAUPRC 比率的箱线图。结果表明,VMPLN 整体表现最佳,尤其是在细胞类型混合水平较高的情况下,其优势更加显著。这表明,与传统的两步法(先聚类后推断基因网络)相比,将网络推断与聚类联合分析可以有效改善网络推断的准确性。
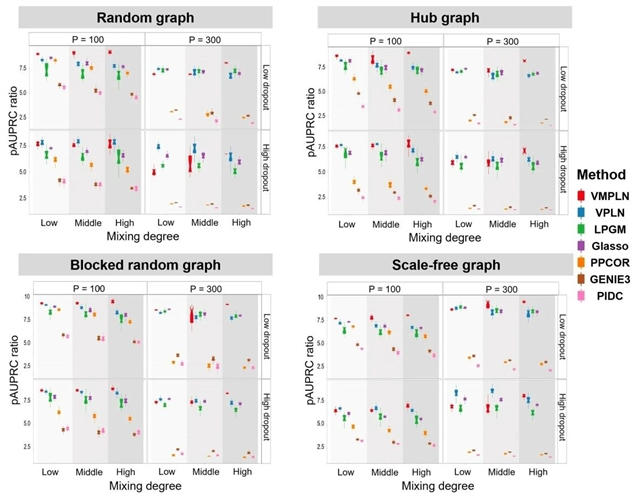
图1. 不同图结构下不同算法的 pAUPRC 比率。参数设置为其默认值或使用其默认方法进行调整。
VMPLN 准确恢复了 scRNA-seq 基准数据中的基因调控关系
研究团队使用两个单细胞RNA测序数据集(Kang数据集和Zheng数据集),对VMPLN与多种现有GRN推断算法进行了效果对比。图2显示了在大多数细胞类型中,VMPLN 实现了最高的部分精确率-召回率(pAUPRC)和早期精确率(early precision),突显出其在联合聚类与网络推断分析中的显著优势。此外,VMPLN 在网络稳定性上也表现出了较高的鲁棒性,为单细胞基因调控网络推断提供了可靠且高效的新工具。
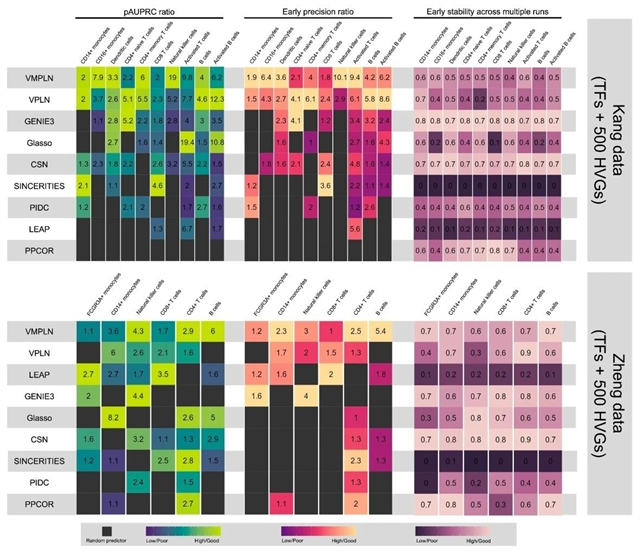
图2. 网络推断算法在两个评估 scRNA-seq 数据集中的表现。颜色代表每种细胞类型中这些指标的缩放值,实际值标记在框中。框中的黑色:随机预测器表现更好。
VMPLN 在 COVID-19 患者 scRNA-seq 数据中揭示关键生物学过程
在 COVID-19 相关研究中,VMPLN 被应用于分析感染 SARS-CoV-2 的患者支气管肺泡灌洗液中的巨噬细胞单细胞 RNA 测序数据。图3 的结果揭示了中度和重度感染患者免疫细胞基因调控网络的显著差异。尤其是在肺泡巨噬细胞中,发现 NR4A1 的靶基因在重度患者中显著上调,并与内质网应激和未折叠蛋白反应等免疫相关过程密切相关。这些发现表明,NR4A1 可能在调控 SARS-CoV-2 感染相关的细胞反应中起关键作用,其相关靶基因(如 IRF1 和 HSP90)可能成为潜在的治疗靶点。
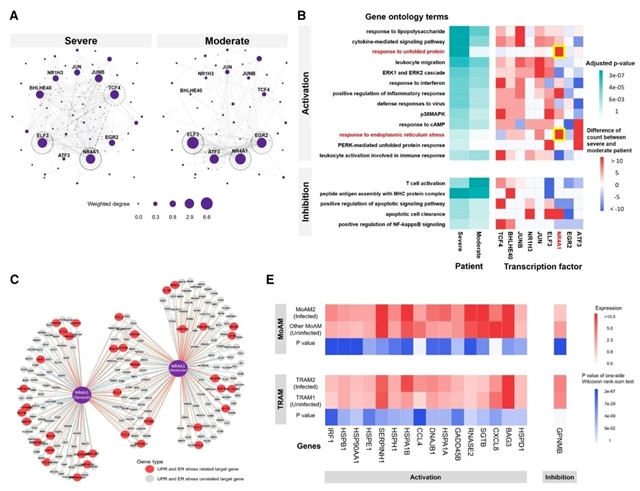
图3. COVID-19 数据的 GRN 分析。(A)重症和中度患者第 4 组巨噬细胞推断的基因调控网络。(B)转录因子靶基因的基因本体富集分析。选择的转录因子为重症和中度患者之间加权度差异较大(>0.2)的转录因子。左图:基因本体术语的 p 值。右图:重症和中度患者基因本体术语中基因数量的差异。(C)NR4A1 调控的基因。(E)巨噬细胞单细胞数据中感染和未感染 SARS-CoV-2 的细胞之间的差异基因表达。
总结与未来方向
本文提出的 VMPLN 方法通过联合聚类与网络推断,为单细胞 RNA 测序(scRNA-seq)数据中的基因调控网络(GRN)推断提供了一种高效且精准的工具。与传统的两步法相比,VMPLN 尤其适用于细胞类型高度混杂的复杂场景。然而,该方法假设调控关系为线性,在强非线性调控机制下可能存在一定的局限性。此外,目前 VMPLN 主要针对 scRNA-seq 数据,未来可以通过整合多组学数据进一步提高方法的灵敏度与准确性。针对细胞类型数量的优化推断仍是一个挑战,可通过引入先验知识或采用更灵活的模型(如 Dirichlet 过程混合模型)进行改进。这些探索为 VMPLN 在多领域中的应用和拓展提供了重要的研究方向。
QB期刊介绍
Quantitative Biology (QB)期刊是由清华大学、北京大学、高教出版社联合创办的全英文学术期刊。QB主要刊登生物信息学、计算生物学、系统生物学、理论生物学和合成生物学的最新研究成果和前沿进展,并为生命科学与计算机、数学、物理等交叉研究领域打造一个学术水平高、可读性强、具有全球影响力的交叉学科期刊品牌。
《前沿》系列英文学术期刊
由教育部主管、高等教育出版社主办的《前沿》(Frontiers)系列英文学术期刊,于2006年正式创刊,以网络版和印刷版向全球发行。系列期刊包括基础科学、生命科学、工程技术和人文社会科学四个主题,是我国覆盖学科最广泛的英文学术期刊群,其中12种被SCI收录,其他也被A&HCI、Ei、MEDLINE或相应学科国际权威检索系统收录,具有一定的国际学术影响力。系列期刊采用在线优先出版方式,保证文章以最快速度发表。
中国学术前沿期刊网
http://journal.hep.com.cn

特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。