短串联重复序列(short tandem repeats, STRs)是一类由1-6个碱基对序列首尾相连、串联重复排列而成的一段DNA序列。由于DNA复制过程中聚合酶滑移等机制,STR具有远高于其他变异类型的突变率,可在极短进化时间尺度上积累丰富的等位基因多态性,从而影响基因调控、表型多样性与物种适应性。尽管STR在遗传变异和复杂性状形成中具有重要作用,它们在大多数的正向遗传关联研究中仍被忽略,特别是STRs对“缺失遗传力”(missing heritability)的潜在贡献,迄今仍不清楚。
中国科学院植物研究所郭亚龙研究组通过对7个独立的拟南芥突变累积(Mutation Accumulation, MA)系进行分析,量化了拟南芥STR的突变率为 5.55×10-3 每代每位点,显著高于单核苷酸多态(SNP)和短片段插入缺失的突变速率。这一结果表明STR在生物进化过程中可能具有重要的作用。在此基础上,研究人员利用高质量组装的基因组,对种间与种内STR的变异模式进行了系统分析,揭示了其变异的特点与规律。进一步结合全球分布的1168份拟南芥自然群体基因组数据,研究人员深入评估了STR长度变异对基因表达、可变剪接及表型的影响,并识别出一批显著关联的STR位点。最终,研究聚焦于STR对遗传力的贡献,发现STR对表达、剪接与表型变异的遗传力贡献分别为0.111、0.143与 0.101,表明STRs在复杂性状的遗传调控中具有不可忽视的重要作用。
基于多维组学数据的整合分析,该研究系统呈现了拟南芥STR的突变图谱、进化动态及其功能影响,为理解STR在复杂性状遗传中的作用提供了新的视角,在一定程度上能够解释“遗传力缺失”之谜。这一成果不仅加深了我们对STR进化机制的认识,也为复杂性状的遗传解析与精准育种提供了新的思路与方法。
该研究成果于8月12日发表于国际学术期刊Genome Biology。植物所博士研究生张稚钦与已毕业博士研究生江娟为共同第一作者,郭亚龙研究员为通讯作者;澳大利亚蒙纳士大学 Sureshkumar Balasubramanian 教授、Sridevi Sureshkumar 教授及 Craig Dent 博士参与研究。该研究得到国家自然科学基金委与国家重点研发计划项目资助。
论文链接:https://doi.org/10.1186/s13059-025-03720-5
(植物多样性与特色经济作物全国重点实验室供稿)
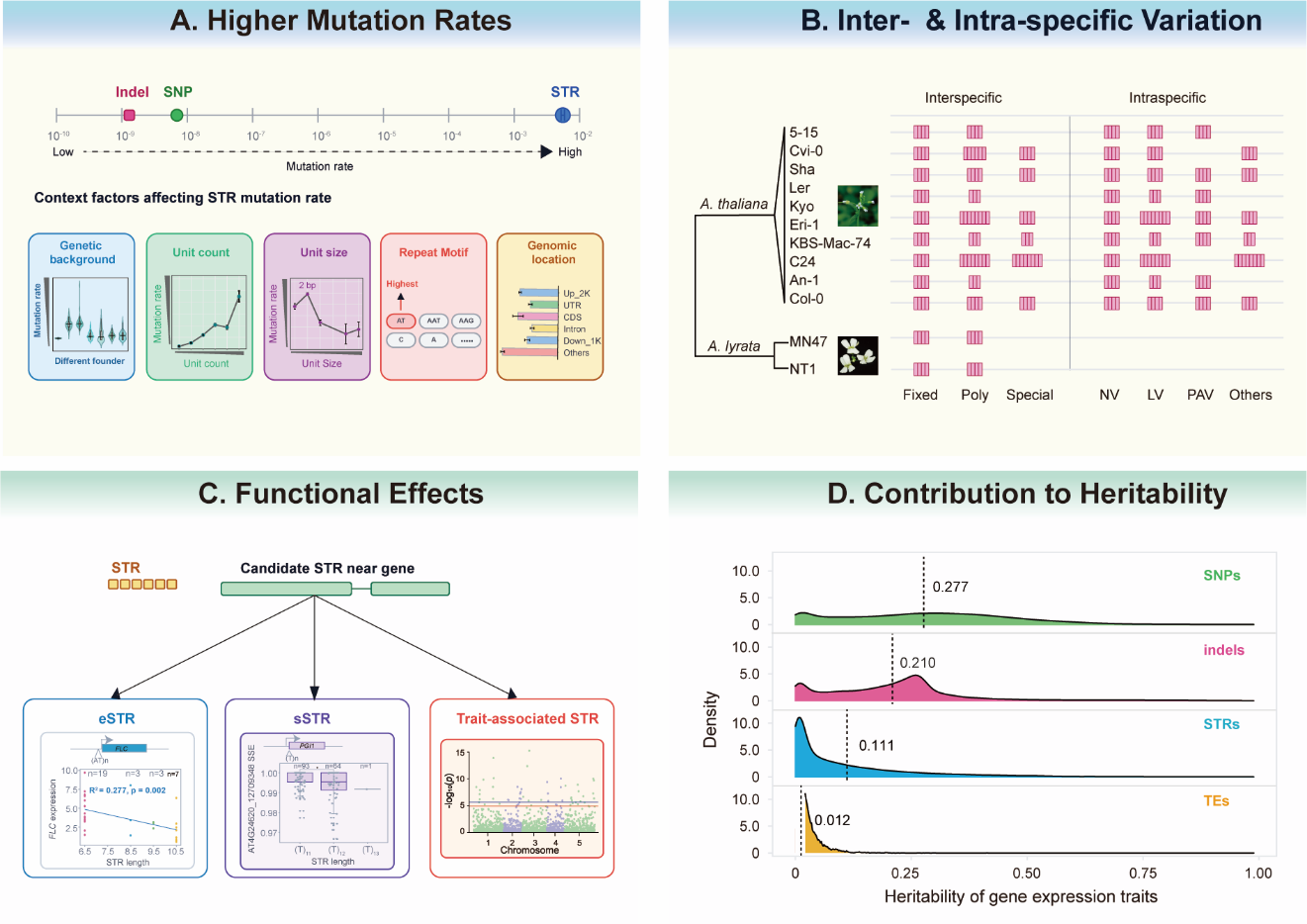 STR 的突变—演化—功能效应。A. STR 的突变速率及其影响因素;B. STR的种间种内变异模式;C. STR与表达、剪接及表型变异的关联;D. STR对表达变异遗传力的贡献。
STR 的突变—演化—功能效应。A. STR 的突变速率及其影响因素;B. STR的种间种内变异模式;C. STR与表达、剪接及表型变异的关联;D. STR对表达变异遗传力的贡献。
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。