|
|
|
|
|
学者提出新型蛋白质适应度预测模型 |
|
实现酶进化中多位点高效重组 |
华南理工大学教授韩双艳团队提出了一种具有自主知识产权的新型蛋白质适应度预测模型——scut-ProFP,并利用该模型成功设计和实现了果胶裂解酶的17位点重组迭代,最优突变体较野生型在高温下热稳定性提升67倍。近日,相关成果分别发表于《蛋白质科学》和《国际生物大分子杂志》。
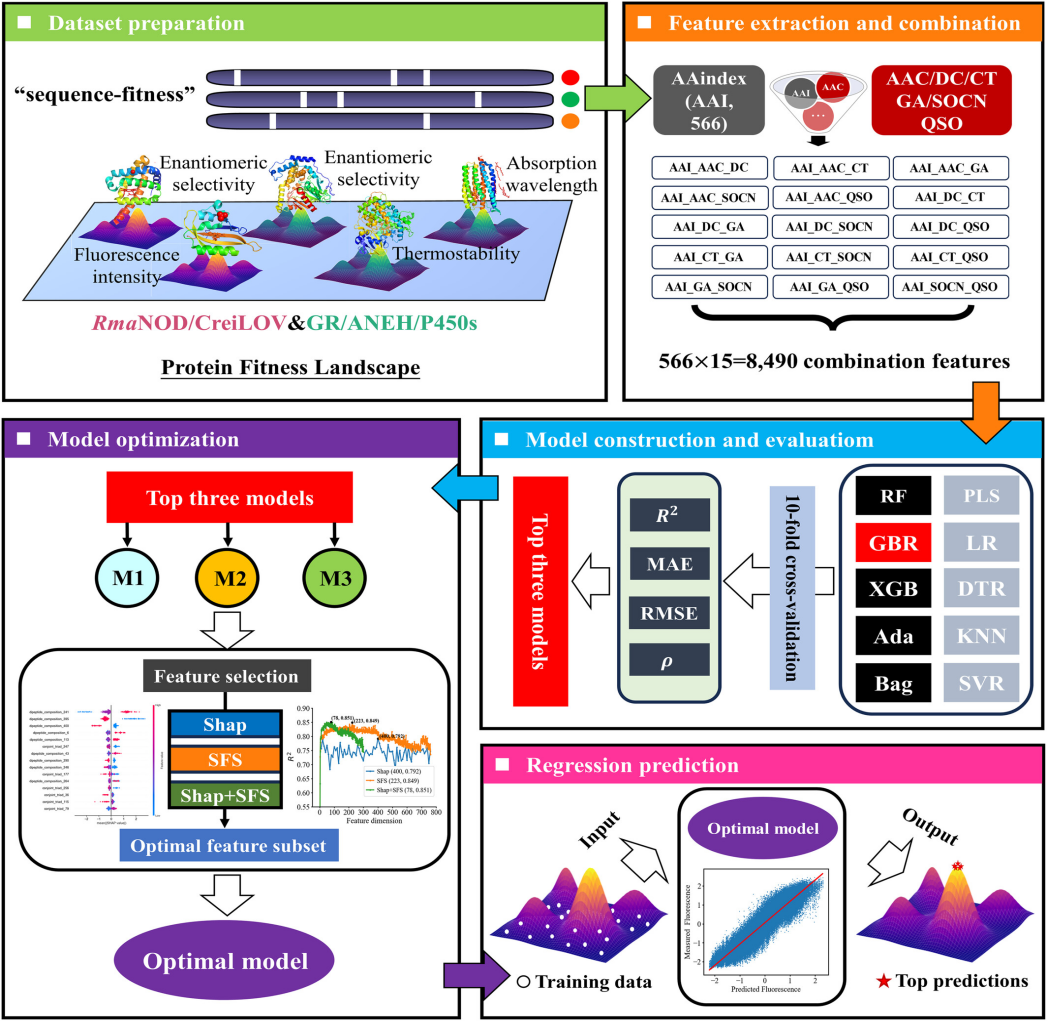 scut-ProFP蛋白质适应度预测模型工作示意图。研究团队供图
scut-ProFP蛋白质适应度预测模型工作示意图。研究团队供图
?
蛋白质氨基酸序列与其功能之间的映射关系可用蛋白质适应度地形表示,寻找解析适应度地形上最高点所对应的氨基酸序列则可能获得进化最优的蛋白,这也是蛋白质改造的目标。然而,多位点变异组合后导致可能的序列数量非常庞大,传统的方法(如定向进化)难以全面探索,容易陷入局部最优。近年来,数据驱动的机器学习为探索适应度地形提供了新的可能。但如何从有限的蛋白质序列中提取关键特征,以优化模型性能,仍是一个亟待解决的问题。
在该研究中,研究人员在国家重点研发计划等项目的资助下,提出了一种具有自主知识产权的命名为scut-ProFP的蛋白质适应度预测模型。该模型通过特征组合全面整合序列信息,并结合高效特征选择算法优化模型性能,实现“序列-功能”精准映射。与现有方法(如PyPEF、PySAR,以及深度学习模型ECNet、EVmutation和UniRep)相比,scut-ProFP在多项指标上均表现卓越,并能够有效捕捉突变位点间复杂的上位性效应,避免遗漏关键突变组合,同时可以从低阶突变体泛化到高阶突变体。
研究人员将scut-ProFP应用于真实的蛋白质工程任务,聚焦酶进化中多位点高效重组关键难题,以果胶裂解酶PMGL-Ba为研究对象,利用scut-ProFP指导了该酶的17位点组合突变。在仅拥有152个样本(约占整个序列空间的0.08%)的情况下,通过两轮迭代,探索了组合序列空间中约20万种可能的突变体,最终获得60个具有卓越热稳定性的组合突变体,设计成功率达100%。其中,最佳突变体P36在75℃和80℃下的半衰期分别提高了67倍和39倍,同时突变体活性提高了2.1倍。
论文通讯作者韩双艳表示,scut-ProFP在小样本条件下能够高效识别高适应度突变体,显著减少湿实验工作量,尤其在多位点组合突变优化方面,scut-ProFP不仅克服了传统方法在高维突变空间中的“组合爆炸”问题,还能有效捕捉突变间复杂的上位性效应,从而实现更优异的突变体设计,为机器学习指导蛋白质进化与改造提供了一种新途径。
相关论文信息:https://doi.org/10.1002/pro.5211
https://doi.org/10.1016/j.ijbiomac.2024.134530
版权声明:凡本网注明“来源:中国科学报、科学网、科学新闻杂志”的所有作品,网站转载,请在正文上方注明来源和作者,且不得对内容作实质性改动;微信公众号、头条号等新媒体平台,转载请联系授权。邮箱:shouquan@stimes.cn。