中山大学中山医学院教授王金凯团队在国家自然科学基金等项目的资助下,开发一种深度学习算法揭示了N6-腺苷酸甲基化(m6A)在核糖核酸(RNA)异构体上的复杂性。近日,相关成果在线发表于《分子细胞》(Molecular Cell)。
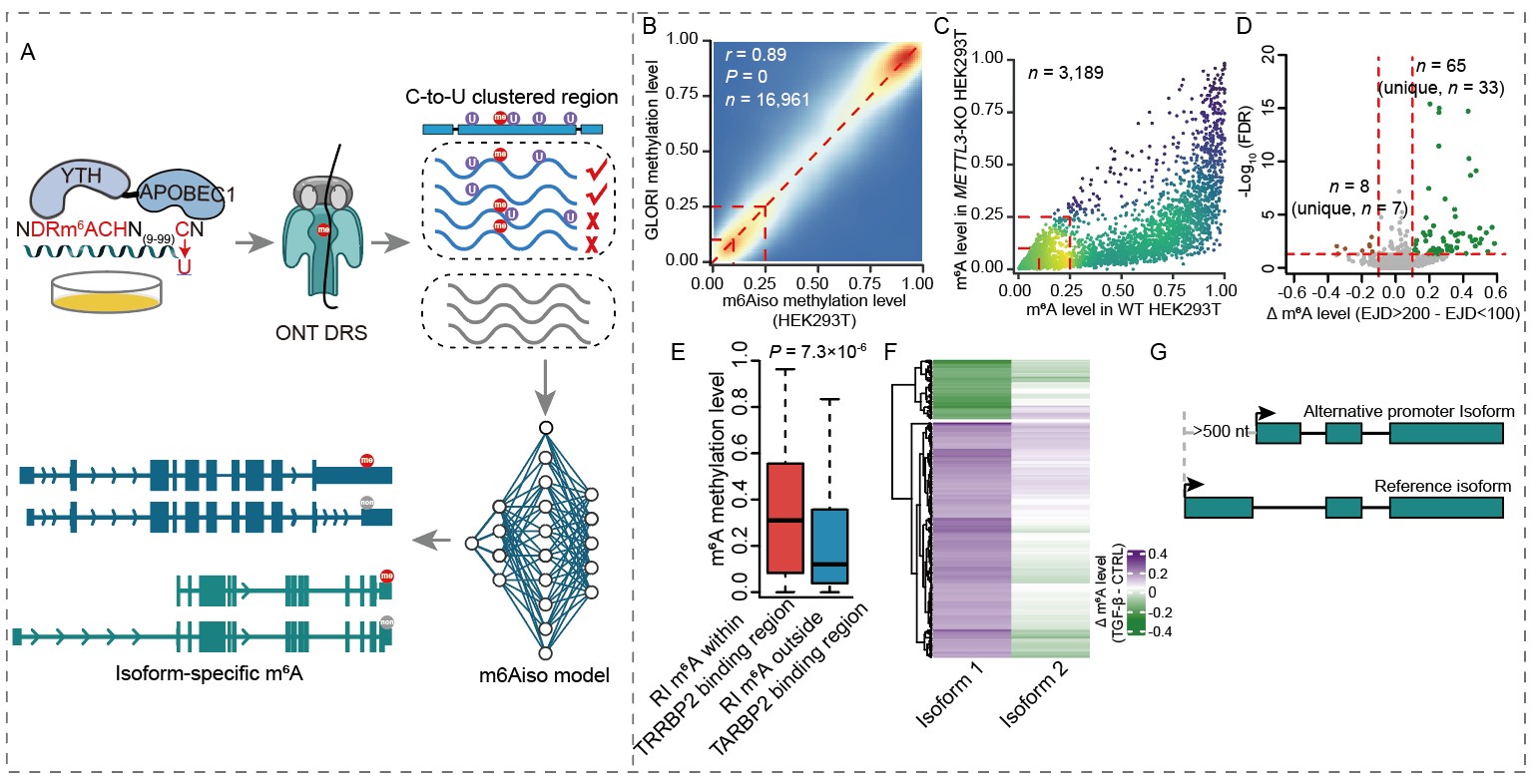 基于内源性标记建立在单分子水平检测m6A的深度学习模型,解析m6A在RNA异构体上的特异性修饰模式和机理。研究团队供图
基于内源性标记建立在单分子水平检测m6A的深度学习模型,解析m6A在RNA异构体上的特异性修饰模式和机理。研究团队供图
?
真核生物的pre-mRNA能够通过选择性加工形成不同的RNA异构体,这些可变的加工方式包括可变剪接、可变加尾。此外,相同基因也存在可变启动子,从而产生具有不同5’端的RNA异构体。这些种类复杂的RNA异构体在哺乳动物细胞中广泛存在,然而m6A是否能够选择性地标记这些RNA异构体并不清楚,尤其是不同RNA异构体上的相同m6A位点是否也能够产生差异的m6A修饰?对这一科学问题理解的欠缺主要是由于技术的匮乏。
研究人员通过APOBEC1-YTH在细胞内使内源的RNA的m6A附近产生C-to-U突变并进行第三代牛津纳米孔直接RNA测序,从而通过已知m6A位点附近10~100 nt的C-to-U突变在单分子水平对m6A进行内源性标记,通过半监督学习进行数据清洗后,获得约一百万个单分子水平的5-mer的m6A电信号,训练出一个在单分子水平准确检测m6A修饰的深度学习模型m6Aiso。
研究人员进而揭示了相同的m6A位点在不同的异构体上也能够通过至少三种不同的机制产生广泛的差异。尤其是在上皮间质转化模型中,TGF-β诱导后,活化的转录因子SMAD3会通过招募METTL3/METTL14/WTAP选择性地促进以SMAD3为启动子的下游RNA的m6A修饰,从而造成相同基因使用不同启动子的异构体呈现选择性的m6A上调。
该研究为理解m6A在异构体上的复杂性以及m6A的选择性调控机理提供了新的视角。
相关论文信息:https://doi.org/10.1016/j.molcel.2025.01.014
版权声明:凡本网注明“来源:中国科学报、科学网、科学新闻杂志”的所有作品,网站转载,请在正文上方注明来源和作者,且不得对内容作实质性改动;微信公众号、头条号等新媒体平台,转载请联系授权。邮箱:shouquan@stimes.cn。